Endokrynologia Pediatryczna, Tom 6 | Rok 2007 | Nr 1(18)
DOI: 10.18544/EP-01.06.01.0018
Genetyczne uwarunkowania autoimmunologicznych chorób tarczycy
Katedra i Klinika Pediatrii i Endokrynologii Akademii Medycznej w Warszawie
Słowa kluczowe: tarczyca, autoimmunologiczne choroby, genetyka
Streszczenie
Choroby autoimmunologiczne stanowią w krajach rozwiniętych najczęstszą przyczynę dysfunkcji tarczycy. Ich rozwój jest wypadkową działania predyspozycji genetycznej oraz czynników środowiskowych. Według modelu statystycznego, opartego na badaniach zgodności zachorowań u bliźniąt monozygotycznych i dizygotycznych, predyspozycja genetyczna jest czynnikiem dominującym w patogenezie autoimmunologicznych zapaleń tarczycy. W pracy przedstawiono aktualne strategie poszukiwania genów podatności obowiązujące w badaniach genetycznych, wyniki badań genów kandydujących oraz wyniki poszukiwania nieznanych genów podatności poprzez skanowanie genomu. Opisano także najnowszą teorię uzasadniającą szczególną predyspozycję płci żeńskiej do autoimmunologicznych chorób tarczycy.
Wstęp
Choroby autoimmunologiczne są najczęstszą przyczyną dysfunkcji tarczycy w krajach rozwiniętych. Jest to dość różnorodna klinicznie grupa chorób, obejmująca chorobę Gravesa-Basedowa, przewlekłe limfocytarne zapalenie tarczycy typu Hashimoto, atroficzne zapalenie tarczycy oraz wiele innych wariantów, jak np. poporodowe zapalenie tarczycy. Należą one do narządowo swoistych chorób autoimmunologicznych. Ich rozwój jest wypadkową działania predyspozycji genetycznej oraz czynników pozagenetycznych zwanych umownie środowiskowymi. Autoimmunologiczne choroby tarczycy z punktu widzenia genetyki należą do chorób wieloczynnikowych, co oznacza, że w patogenezie tych chorób bierze udział wiele genów o różnych loci i zróżnicowanej penetracji. Dodatkowo może występować heterogeniczność genetyczna, tzn. różne geny mogą wywoływać ten sam fenotyp choroby. Choroby wieloczynnikowe nie mogą być analizowane według praw dziedziczenia mendlowskiego. Ograniczone znaczenie ma analiza rodowodów i utrudnione jest mapowanie genów podatności. Niektóre z nich zidentyfikowano, wśród nich są geny dające ogólną podatność na choroby autoimmunologiczne, a także takie, które są sprzężone z określoną jednostką chorobową.
Epidemiologia genetyczna autoimmunologicznych chorób tarczycy
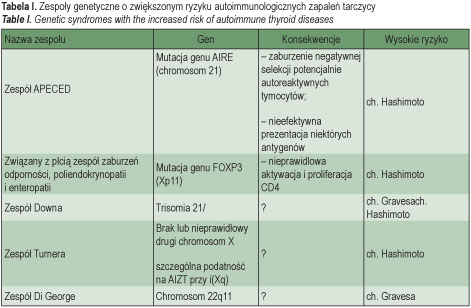
O istotnym udziale czynników genetycznych w patogenezie autoimmunologicznych zapaleń tarczycy (AIZT) świadczą dane epidemiologiczne. Rodzinne występowanie choroby Gravesa było znane od ponad 100 lat. Potwierdziły to badania oparte na porównaniu częstości zachorowań w rodzinach obciążonych ryzykiem z częstością w zdrowej populacji. Występowanie rodzinne przeciwciał przeciwtarczycowych u rodzeństwa chorych z AIZT opisano już w latach sześćdziesiątych XX wieku. Udowodniły to liczne później publikowane prace. Na ich podstawie stwierdzono autosomalnie dominujące dziedziczenie skłonności do produkcji przeciwciał przeciwtarczycowych [1, 2]. Mocnym dowodem udziału czynników genetycznych w patogenezie autoimmunologicznych zapaleń tarczycy są zespoły genetyczne z wysokim ryzykiem ich występowania (Tabela I).
W badaniach par bliźniąt stwierdza się większą zgodność zachorowań na chorobę Gravesa i chorobę Hashimoto u bliźniąt monozygotycznych niż dizygotycznych [3, 4]. Według przedstawionego przez Brixa i wsp. modelu statystycznego, opartego na badaniach zgodności zachorowań u bliźniąt monozygotycznych i dizygotycznych, predyspozycja genetyczna jest czynnikiem dominującym w patogenezie autoimmunologicznych zapaleń tarczycy. Autorzy podają, że czynniki genetyczne stanowią 79%, a środowiskowe tylko 21% udziału w patogenezie AIZT [4]. Wobec tak istotnej przewagi czynników genetycznych wydaje się, że poznanie ich stworzyłoby nowe możliwości wyjaśnienia przyczyn powstania i rozwoju choroby.
Podstawowe metody poszukiwania genów podatności
W chorobach wieloczynnikowych strategia badań genetycznych opiera się na poszukiwaniu sprzężeń i asocjacji genów kandydujących oraz na poszukiwaniu nieznanych genów związanych z chorobą poprzez skanowanie całego genomu.
Analiza sprzężeń właściwie odnosi się do odległości genetycznej między loci genów.
Podstawą tej metody jest założenie, że geny położone na chromosomie blisko siebie w czasie mejozy kosegregują i prawdopodobieństwo ich rekombinacji jest niskie. Jeżeli więc badany marker położony jest w sąsiedztwie genu odpowiadającego za rozwój choroby, to będzie kosegregował z chorobą w obciążonych rodzinach. Miarą prawdopodobieństwa sprzężenia jest logarytm szans faworyzujących sprzężenie, czyli logarytm szans (Logarithm of the odds, LOD). Termin ten oznacza logarytm stosunku prawdopodobieństwa sprzężenia do prawdopodobieństwa braku sprzężenia. Klasyczny test sprzężeń jest oceną parametryczną. W analizie chorób wieloczynnikowych bardziej przydatny jest test nieparametryczny. Przykładem takiej analizy jest badanie par rodzeństw dotkniętych chorobą. Określa się występowanie u nich wspólnych genów w loci markerowych. Większa niż spodziewana zgodność proporcji alleli u chorego rodzeństwa sugeruje, że rejon zawierający badany marker zawiera gen choroby. Lander i Kruglyak stworzyli schemat badań sprzężeń genetycznych w chorobach wieloczynnikowych. Według ich sugestii w ocenie parametrycznej LOD większy niż 3,3 wskazuje na istotne sprzężenie. Dla oceny nieparametrycznej bariera jest wyższa. Sprzężenie można traktować jako pewne tylko wtedy, kiedy jest potwierdzone w dwóch niezależnych próbach. Wartość LOD niższa niż –2 wyklucza sprzężenie [5].
Techniki badania sprzężeń są wartościowym narzędziem w analizie genów warunkujących choroby wieloczynnikowe, bo wykrywają geny, które mają największy wpływ na rozwój tych chorób. Metoda ta jednak ma swoje ograniczenia, np. nie wykrywa genów o mniejszym wpływie i nie nadaje się do mapowania mniejszych rejonów genetycznych. Bardziej czułe i lepsze w mapowaniu określonych rejonów jest badanie asocjacji. Termin ten określa zjawisko współwystępowania alleli i/ lub fenotypów. Analiza asocjacji jest wysoce swoista i może wykrywać geny biorące udział tylko w 5% ogólnej genetycznej kontrybucji choroby. Klasyczną analizę asocjacji prowadzi się w grupie niespokrewnionych chorych i etnicznie dobranych niespokrewnionych zdrowych kontrolach. Jeżeli dany allel występuje istotnie częściej u pacjentów niż w kontroli, jest to dowodem jego asocjacji z chorobą. Prawdopodobieństwo choroby u osobnika posiadającego dany allel w stosunku do osobnika nieposiadającego tego allela jest wyrażone jako współczynnik względnego ryzyka (Relative Risk, RR) . Ograniczeniem tej metody jest konieczność starannego dobrania grupy kontrolnej, inaczej wynik może być obciążony dużym błędem. Najbardziej wartościowe są testy asocjacji oparte na badaniu rodzin, gdzie tworzy się wewnętrzne grupy kontrolne. Przykładem takiej analizy jest test nierównowagi transmisji (Transmission disequilibrium test, TDT). Opiera się on na analizie markerowych alleli rodziców, które są transmitowane i nietransmitowane do chorego potomstwa. Porównanie częstości występowania poszczególnych alleli w grupie transmitowanej i w grupie nietransmitowanej pozwala na wyodrębnienie alleli związanych z chorobą.
Wykrycie sprzężeń w danym rejonie genetycznym nie jest jednak wystarczającym dowodem na istotny udział danego genu w patogenezie choroby. Tomer i Davies proponują algorytm wykorzystany w wielu opracowaniach genetycznych, pozwalający na uznanie badanego loci za gen podatności [6]. Obejmuje on pięć kolejnych etapów badań:
1. Identyfikacja sprzężonego locus – skanowanie całego genomu z użyciem markerów mikrosatelitarnych.
2. Potwierdzenie sprzężenia w danym rejonie – w dwóch niezależnych próbach.
3. Mapowanie sprzężonego locus – ograniczenie badanego rejonu do kilkuset kilozasad poprzez analizę asocjacji znanych markerów.
4. Badanie genów w rejonie sprzężeń – identyfikacja polimorfizmów pojedynczych zasad (S ingle nucleotide polymorphisms – SNPs) w obrębie genów w rejonie sprzężenia i potwierdzenie ich asocjacji z chorobą.
5. Badania czynnościowe – wykazanie, że dany polimorfizm zmienia funkcję genu w sposób zwiększający ryzyko choroby.
Analiza sprzężeń i asocjacji genów kandydujących
Badanie genów kandydujących obejmuje badanie markerów polimorficznych w obrębie poszczególnych genów, które zostały wytypowane w związku z tym, że defekt ich funkcji daje skutek w postaci choroby. Idealny gen kandydujący do roli genu podatności to gen o znanej sekwencji i lokalizacji, który poprzez swoją fizjologiczną funkcję gra istotną rolę w patogenezie choroby.
Zgodnie z takim założeniem przedmiotem zainteresowania w AIZT powinny być geny czynników biorących udział w inicjowaniu i regulacji odpowiedzi immunologicznej, geny antygenów swoistych dla tarczycy, mających kluczowe znaczenie w patogenezie choroby oraz geny lub rejony genetyczne wybrane na podstawie grup ryzyka (patrz Tabela I).
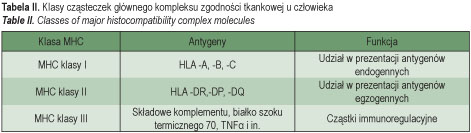
Idealnym kandydatem z uwagi na znaczny polimorfizm i kodowanie wielu genów związanych z odpowiedzią immunologiczną był rejon głównego kompleksu zgodności tkankowej (MHC), zlokalizowany na krótkim ramieniu chromosomu 6 (6p21). Obejmuje on wiele genów odznaczających się największym polimorfizmem z dotąd poznanych. Mają one znaczenie zarówno w inicjacji, jak i fazie efektorowej odpowiedzi immunologicznej. Koduje on trzy klasy cząsteczek (Tabela II). Cząstki MHC klasy I i II grają główną rolę w inicjacji adaptacyjnej odpowiedzi immunologicznej. Antygeny peptydowe mogą być rozpoznane przez receptor limfocytu T tylko wtedy, gdy są prezentowane razem z cząstką MHC na powierzchni komórki prezentującej antygen.
W chorobie Gravesa-Basedowa początkowo wykazano asocjację rejonu HLA-B8, a następnie silniejszą asocjację z HLA-DR3. Rejony te pozostają z sobą w silnej nierównowadze sprzężeń. Wiele kolejnych analiz potwierdziło związek HLA-DR3 i choroby Gravesa-Basedowa u rasy białej [6, 7]. Według danych Segni i wsp. istnieje także preferencja transmisji odojcowskiej alleli podatności [8]. W populacjach innych niż kaukaska rejony sprzężeń wykazywały znacznie większe zróżnicowanie. W populacji japońskiej wykazano sprzężenie w rejonie HLA-B35, -B46, -A2, -DPB1*0501 [9–11], w chińskiej HLA -B46, -DR9, -DRB1*303, -DQB1*0303 [12], a u rasy czarnej w Afryce Południowej HLA-DR1 i -DR3 [13].
Badano także asocjację HLA i cech klinicznych choroby Gravesa-Basedowa. Wykazywano asocjację oftalmopatii z HLA-B8 i DR3 [14], allotyp ten wiązał się także z wysokim ryzykiem nawrotu po leczeniu tyreostatykami [15] oraz opornością na leczenie jodem radioaktywnym [16]. Obserwacji tych nie udało się potwierdzić w kolejnych niezależnych próbach. Podobnie niepotwierdzone jest protekcyjne działanie allotypu HLA-DR4 oraz HLA-DR7 [6,17].
W zapaleniu tarczycy typu Hashimoto z wolem oraz w poporodowym zapaleniu tarczycy wykazano słabą asocjację z allotypem HLA-DR4, -DR5 u rasy białej, a w innych populacjach HLA-Drw53 i HLA-DR9. Natomiast atroficzne zapalenie tarczycy wykazywało asocjację z HLA-DR3 [6, 17].
Kolejnym genem kandydującym o istotnym znaczeniu jest gen CTLA-4 (cytotoxic T lymphocyte antigen 4). Jest on negatywnym regulatorem pobudzenia limfocytu T, pojawiającym się na jego powierzchni po pobudzeniu. Jest kluczowym inhibitorem aktywacji limfocytu T. Ludzki gen CTLA-4 jest położony na długim ramieniu chromosomu 2 (2q33). W nieparametrycznej analizie sprzężeń w rodzinach obciążonych chorobą Gravesa stwierdzono silne sprzężenie w rejonie genu CTLA-4. Z dotychczas opisanych polimorfizmów w rejonie CTLA-4, najważniejsze i najlepiej zbadane są trzy: zamiana A na G w pozycji 49 eksonu 1 (A/G 49); zamiana C na T w pozycji -318 (C/T -318) w rejonie promotorowym genu oraz polimorfizm markera mikrosatelitarnego w nieulegającym transkrypcji rejonie 3’ exonu 4 (3’ UTR) CTLA-4. W chorobie Gravesa-Basedowa wykazano asocjację polimorfizmu A/G 49 oraz 3’UTR mikrosatelity niezależnie od pochodzenia etnicznego [18, 19, 20]. Wynik ten potwierdzono także w teście nierównowagi transmisji. Stwierdzono, że obydwa ww. polimorfizmy występują częściej także u pacjentów z zapaleniem tarczycy typu Hashimoto [21, 22]. Rejon CTLA-4 wykazuje także asocjację ze skłonnością do produkcji przeciwciał przeciwtarczycowych [23] oraz z przebiegiem klinicznym choroby. Według tych badań polimorfizm A/G 49 zmniejsza szansę na uzyskanie remisji po tyreostatykach i ma wpływ na cięższy przebieg tyreotoksykozy u pacjentów z chorobą Gravesa [24]. Nie wszystkim autorom udało się to jednak potwierdzić. Polimorfizm A/G 49 genu CTLA-4 jest związany z ogólną podatnością na choroby autoimmunologiczne i wykazuje asocjację nie tylko z chorobami tarczycy, ale także m.in. z cukrzycą typu I.
Wyniki analizy innych genów związanych z odpowiedzią immunologiczną przedstawiono w tabeli III.

Spośród genów swoistych dla tkanki tarczycowej badanych w AITD największe nadzieje budzi gen tyreoglobuliny. Silne sprzężenie z AIZT w rejonie genu tyreoglobuliny na chromosomie 8q24 wykryto w dwóch niezależnych badaniach skanujących genom [25, 26]. Dodatkowo udowodniono sprzężenie z AIZT dwóch markerów mikrosatelitarnych położonych w obrębie genu tyreoglobuliny (Tgms1 i Tgms2) [26]. Potwierdzono także asocjację Tgms2 zarówno w klasycznej analizie, jak i w TDT. Istnieje jednak możliwość, że zależność ta jest wynikiem działania nieznanego genu w tym locus pozostającego w nierównowadze sprzężeń z genem tyreoglobuliny [6]. Inne geny swoiste dla tarczycy badane jako geny podatności na AIZT to gen receptora TSH (chromosom 14q31), gen peroksydazy tarczycowej (chromosom 2p25). Wyniki analiz nie potwierdziły znaczenia tych genów jako istotnych genów podatności [6, 17].
Poszukiwanie nowych rejonów sprzężeń przez skanowanie całego genomu
Aby analizować sprzężenia całego genomu w przypadku chorób wieloczynnikowych, należy dysponować odpowiednio dużą grupą rodzin z więcej niż jednym członkiem rodziny dotkniętym chorobą oraz mapą wysoce polimorficznych, blisko położonych markerów pokrywających cały genom. Najczęściej do screeningu całego genomu używa się markerów mikrosatelitarnych. Są to rejony genomu złożone z powtórzeń krótkich sekwencji, najczęściej dwuzasadowe powtórzenia CA. Zwykle nie pełnią one żadnej znanej funkcji. Są wysoce polimorficzne z uwagi na liczbę powtórzeń u poszczególnych osobników i jednorodnie rozmieszczone w genomie w odległości co najwyżej 1 mln par zasad.
Opublikowane w ostatnich latach trzy największe badania całego genomu pochodzą z Japonii, Stanów Zjednoczonych i Anglii. W badaniu japońskim uczestniczyły 123 rodziny [25]. Znaleziono sprzężenie w rejonie genu cytokin: 5q31, dla którego maksymalna wartość LOD (maximal LOD score, MLS) wynosiła 3,14, oraz w rejonie genu tyreoglobuliny 8q24, który wykazywał silniejsze sprzężenie z chorobą Hashimoto (MLS=3,77 ) i nieco słabsze z różnymi postaciami AIZT (MLS=2,31).
W badaniu amerykańskim wzięły udział 102 rodziny [6]. Znaleziono razem 7 loci: trzy wykazujące sprzężenie z różnymi postaciami AIZT: 6p (MLS=2,0), 8q (MLS=3,5), 10q (MLS=4,1, trzy loci pozostające w sprzężeniu z chorobą Gravesa: 7q (MLS=2,3), 14q (MLS=2,1), 20q (MLS=3,3) oraz jedno loci sprzężone tylko z chorobą Hashimoto: 12q (MLS=3,4).
Badanie angielskie obejmowało największą grupę pacjentów, analizowało 558 rodzin z AIZT [27]. Znaleziono sprzężenie z chorobą Gravesa-Basedowa w rejonie 18p11 (MLS=2,5), 2q36 (MLS=2,2) oraz 11p15 (MLS=2,0).
Genetyczne uzasadnienie większej zachorowalności na AIZT u płci żeńskiej
Wszystkie postaci autoimmunologicznych chorób tarczycy występują u kobiet 5-10 razy częściej niż u mężczyzn, co sugeruje wpływ steroidów płciowych na rozwój choroby.
Jak udowodniono w licznych eksperymentach, obserwowane różnice w przebiegu chorób autoimmunologicznych u płci żeńskiej i męskiej wykraczają daleko poza różnice hormonalne. Według ostatniej koncepcji potencjalnym mechanizmem, poprzez który może dojść do zaburzenia autotolerancji, jest ukierunkowana inaktywacja chromosomu X.
Osobniki żeńskie są mozaikami dwóch linii komórkowych: jedna linia posiada odojcowski X, druga odmatczyny jako aktywny materiał genetyczny. Zwykle we wszystkich tkankach stosunek tych linii wynosi w przybliżeniu 50:50. Zjawisko ukierunkowanej inaktywacji zachodzi wówczas, gdy w różnych tkankach większa część komórek inaktywuje ten sam chromosom X (np 80% lub więcej). W takiej sytuacji może się zdarzyć, że antygeny własne zakodowane na chromosomie X mogą nie mieć dostatecznej ekspresji w tkankach związanych z indukcją tolerancji immunologicznej (np. grasica), natomiast będą obecne w innych tkankach w wysokiej dawce i będą rozpoznawane jak inne obce antygeny [28, 29]. Dowodem słuszności tej koncepcji jest większa częstość ukierunkowanej inaktywacji chromosomu X u kobiet ze sklerodermą [30] oraz u kobiet z autoimmunologicznym zapaleniem tarczycy [29].
Wnioski
Na podstawie dotychczasowych badań wydaje się, że bardziej efektywne jest badanie genów kandydujących. Wśród nich można uznać, że pewny i powtarzalny jest udział dwóch rejonów: genów układu HLA oraz CTLA-4. Obiecujące wyniki dotyczą genu tyreoglobuliny, ale wymagają potwierdzenia w dalszych badaniach. Wyniki skanowania całego genomu w poszukiwaniu nowych genów podatności pokazują, że nie ma zgodności rejonów sprzężeń w badanych populacjach. Może to wynikać z różnic etnicznych, ale zwraca uwagę fakt, że nie znaleziono sprzężeń w rejonach uznanych za geny podatności niezależne od pochodzenia etnicznego – rejon HLA, CTLA-4, Xp11. Dowodzi to ograniczeń metody wykrywającej jedynie silnie sprzężone loci, a choroby wieloczynnikowe, do których należą autoimmunologiczne zapalenia tarczycy, są wypadkową działania wielu genów o niewielkim wpływie i różnej penetracji. Ich wykrycie wymaga bardziej czułych metod. Jedną z propozycji jest zastąpienie analizy sprzężeń badaniem asocjacji w rozległych rejonach genetycznych [27].
Piśmiennictwo
1. Phillips D., McLachlan S., Stephenson A. et al.; Autosomal dominant transmission of autoantibodies to thyroglobulin and thyroid peroxidase; J. Clin. Endocrinol. Metab. 1990:70, 742-746
2. Phillips D., Prentice L., Upadhyaya M. et al.; Autosomal dominant inheritance of autoantibodies to thyroid peroxidase and thyroglobulin – studies in families not selected for autoimmune thyroid disease; J. Clin. Endocrinol. Metab. 1991:72, 973-975
3. Brix T.H., Kyvik K.O., Hegedius L.; A population-based study of chronic autoimmune hypothyroidism in Danish twins; J. Clin. Endocrinol. Metab. 2000:85, 536-539
4. Brix T.H., Kyvik K.O., Christensen K., Hegedius L.; Evidence for a major role of heredity in Graves’ disease: a population-based study of two Danish twin cohorts; J. Clin. Endocrinol. Metab. 2001:86, 930-934
5. Lander E., Kruglyak L.; Genetic dissection of complex traits: guidelines for interpreting and reporting linkage results; Nat. Genet. 1995:1, 241-247
6. Tomer Y., Davies T.F.; Searching for the autoimmune thyroid disease susceptibility genes: from gene mapping to gene function; Endocrine Rev. 2003:24(5), 694-717
7. Farid N.R., Stone E., Johnson G.; Graves’ disease and HLA: clinical and epidemiologic associations; Clin. Endocrinol. (Oxf) 1980:13, 535-544
8. Segni M., Pani M.A., Pasquino M., Badenhoop K.; Familial clustering of juvenile thyroid autoimmunity: higher risk is conferred by Human Leukocyte Antigen DR3-DQ2 and thyroid peroxidase antibody status in fathers; J. Clin. Endocrinol. Metab. 2002:87(8), 3779-3782
9. Kawa A., Nakamura S., Nakazawa M. et al.; HLA-Bw35 and B5 in Japanese patients with Graves’ disease; Acta Endocrinol. (Copenh.) 1977:86, 754-757
10. Onuma H., Ota M., Sugenoya A. et al.; Association of HLA-DPB1*0501 with early onset Graves’ disease in Japanese; Hum. Immunol. 1994:39, 195-201
11. Dong R.P., Kimura A., Okubo R. et al.; HLA-A and DPB1 loci confer susceptibility to Graves’ disease; Hum. Immunol. 1992:35, 165-172
12. Cavan D.A., Penny M.A., Jacobs K.H. et al.; The HLA association with Graves’ disease is sex – specific in Hong Kong Chinese subjects; Clin. Endocrinol. (Oxf) 1994:40, 63-66
13. Omar M.A., Hammond M.G., Desai R.K. et al.; HLA class I and II antigens in South African blacks with Graves’ disease; Clin. Immunol. Immunopathol. 1990:54, 98-102
14. Kendall-Taylor P., Stephenson A., Stratton A. et al.; Differentiation of autoimmune ophtalmopathy from Graves’ hyperthyroidism by analysis of genetic markers;
15. Allanic H., Fauchet R., Lorcy Y. et al.; A prospective study of the relationship between relapse of hyperthyroid Graves’ disease after antithyroid drugs and HLA haplotype; J. Clin. Endocrinol. Metab. 1983:57, 719-722
16. de Bruin T.W., Bolk J.H., Bussemaker J.K. et al.; Graves’ disease: immunological and immunogenetic indicators of relapse; Br. Med. J. 1988:296, 1292-1295
17. Vaidya B., Kendall-Taylor P., Pearce S.H.; The genetics of autoimmune thyroid disease; J. Clin. Endocrinol. Metab. 2002:87(12), 5385-5397
18. Kouki T., Gardine C.A., Yanagawa T., DeGroot L.J.; Relation of three polymorphisms of the CTLA-4 gene in patients with Graves’ disease; J. Endocrinol. Invest. 2002:25, 208-213
19. Akamizu T., Sale M.M., Rich S.S. et al.; Association of autoimmune thyroid disease with microsatellite markers for the thyrotropin receptor gene and CTLA-4 in Japanese patients; Thyroid 2000:10, 851-858
20. Park Y.J., Chung H.K., Park D.J. et al.; Polymorphism in the promoter and exon 1 of the cytotoxic T lymphocyte antigen-4 gene associated with autoimmune thyroid disease in Koreans; Thyroid 2000:10, 453-459
21. Kotsa K.,Watson P.F., Weetman A.P.; A CTLA-4 gene polymorphism is associated with both Graves’ disease and autoimmune hypothyroidism; Clin. Endocrinol. (Oxf) 1997:46, 551-554
22. Ueda H., Howson J.M.M., Esposito L. et al.; Association of the T-cell regulatory gene CTLA-4 with susceptibility to autoimmune disease; Nature 2003:423, 506-511
23. Tomer Y., Greenberg D.A., Barbesino G. et al.; CTLA-4 and not CD28 is a susceptibility gene for thyroid autoantibody production; J. Clin. Endocrinol. Metab. 2001:86(4), 1687-1693
24. Kinjo Y., Takasu N., Komiya I. et al.; Remission of Graves’ hyperthyroidism and A/G polymorphism at position 49 in exon 1 of cytotoxic T lymphocyte- associated molecule-4 gene; J. Clin. Endocrinol. Metab. 2002:87: 2593-2596
25. Sakai K., Shirasawa S., Ishikawa N. et al.; Identfication of susceptibility loci for autoimmune thyroid disease to 5q31-q33 and Hashimoto’s thyroiditis to 8q23-q24 by multipoint affected sib-pair linkage analysis in Japanese; Hum. Mol. Genet. 2001:10, 1379-1386
26. Tomer Y., Greenberg D.A., Conception E. et al.; Thyroglobulin is a thyroid specific gene for the familial autoimmune thyroid diseases; J. Clin. Endocrinol. Metab. 2002:87, 404-407
27. Taylor J.C., Gough P.J., Hunt T.H. et al.; A genome – wide screen in 1119 relative pairs with autoimmune thyroid disease; J. Clin. Endocrinol. Metab. 2006:91(2), 646-653
28. Davies T.F.; X versus X- the fight for function within the female cell and the development of autoimmune thyroid disease; J. Clin. Endocrinol. Metab. 2005:90(11), 6332-6333
29. Brix T.H., Knudsen G.P., Kristiansen M. et al.; High frequency of skewed X-chromosome inactivation in female with autoimmune thyroid disease: A possible explanation for the female predisposition to thyroid autoimmunity; J. Clin. Endocrinol. Metab. 2005:90(11), 5949-53
30. Ozbalkan Z., Bagislar S., Kiraz S. et al.; Skewed X chromosome inactivation in blood cells of women with scleroderma; Arthritis Rheum 52, 1564-1570
31. Tsurumaru M., Kawasaki E., Hiroaki I. et al.; Evidence for the role of Small Ubiquitin-Like Modifier 4 as a general autoimmunity locus in the Japanese population; J. Clin. Endocrinol. Metab. 2006:91(8), 3138-3143